使用PyTorch炼丹的过程中,我们最怕的就是在DataLoader里debug,原因无他:多进程驱动的DataLoader很难给出清晰的traceback报错,即便将num_worker设为0不启用多进程,有时一个DataLoader Worker PID XXX is killed by signal: Killed或者Segmentation Fault还是能让用户一脸懵逼。
新手炼丹师最常见到的问题就是在DataLoader的__getitem__方法调用cv2读取数据导致的segmentation fault,原因是cv2的多线程读取与DataLoader多进程读取交织会不定期造成死锁;或者是OS共享内存耗尽导致的DataLoader worker killed by signal。多年积赞的经验让笔者自以为在PyTorch调试技巧方面已经修炼的炉火纯青,可惜最近还是在一个困扰PyTorch社区3年的问题上栽了跟头。下面记录一下排查经过:
这篇英文博客定量分析了各种multiprocessing方式(spawn/fork/forkserver)在不同设置下的内存泄露情况,如果你希望深入了解,还请拜读!如果你只希望定性了解解决方案,下面的内容也能满足你的需求。
太长不看版
- 不要在Python多进程setting下使用大的内建对象(list, dict, ...),而应使用有C binding的库提供的数据对象(如numpy.Array,pyarrow.Array);
- 单机多实验进行data loading时,尝试读取存储在
/dev/shm/中的memmap规避在每个实验中重复IO数据集带来的overhead。
起因
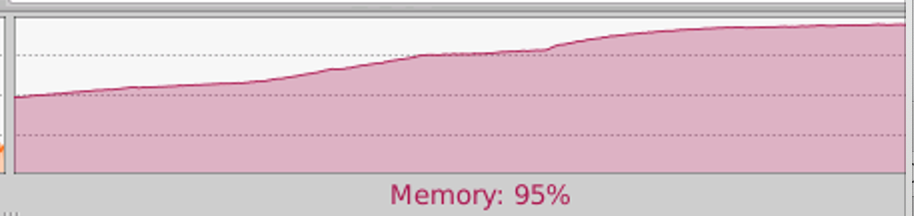
最近在单机开展多个实验时发现,实验数量一多,其中某个实验就会不定期报DataLoader Worker PID XXX is killed by signal: Killed;观察Memory Curve发现内存占用稳定增长,直到每个epoch结束后释放,一旦内存占满就报Killed:
排查
这其实是多进程编程中老生常谈的问题:在基于fork的多进程实现中,每次fork会让子进程得到不同的虚拟空间地址,但此时其映射的还是父进程的物理内存空间,可以让子进程高效率读取父进程的memory数据。一旦子进程有写操作就会触发操作系统的copy-on-write异常,系统会拷贝出另一块空间供子进程使用。
但实际上,除了数据增广,一般没有人会在DataLoader里改写对象吧?笔者上面的例子对大内存对象的唯一的操作是这样一句话:
scan = self.scans[ref['scan_id']] # self.scans反序列化后是一个占用9GB内存的大dict这句话是在一个继承Dataset类的数据集类的__init__方法里。从常识来看,这句话是对self.scans的Access操作,不应该导致copy-on-write。
但经这个issue的commect提醒,我们可以回顾一下PyObject在C中的结构:
[object.h]
/* PyObject_HEAD defines the initial segment of every PyObject. */
#define PyObject_HEAD
_PyObject_HEAD_EXTRA
int ob_refcnt; // 引用计数
struct _typeobject *ob_type;
#define PyObject_VAR_HEAD
PyObject_HEAD
int ob_size; /* Number of items in variable part */每一次在fork后的子进程里对大型Python内建对象的引用,都会增加对象的ob_refcnt,变相write了这个对象,这个操作进而触发copy-on-write也就不奇怪了。
解决方法
Numpy等科学计算库从底层提供了数值、字符串等常用数据的handler np.Array,一个改写的例子如下:
改写前:
class traindataset(Dataset):
def __init__(self):
self.utt2fpath = {'utt1': './t1.wav', 'utt2': './t2.wav' ...}
self.utts = list(self.utt2fpath.keys())
def __getitem__(self, item):
utt = self.utts[item]
fpath = self.utt2fpath[utt]
...改写后:
import numpy as np
class traindataset(Dataset):
def __init__(self):
self.utt2fpath = np.array([['utt1', './t1.wav'], ['utt2', './t2.wav'], ...])
def __getitem__(self, item):
utt = self.utt2fpath[item][0]
fpath = self.utt2fpath[item][1]
...相信不少人都习惯了在Dataset中使用python内建的list存储路径以便在__getitem__中读取,看了本文之后,你完全就可以用np.array避免不必要的memory overhead了。
注意!np.array只支持数值类型及字符串的C-binding,其他的python自建对象仍然以PyObject形式存在并通过pickle/unpickle的方式兼容numpy的逻辑。
如何判断?
>> np.array({'d': 'd'})
array({'d': 'd'}, dtype=object) # dtype为object,代表numpy只是提供了PyObject的Wrapper,无法解决问题
这里也给其他研究者提一个醒,回到我遇到的scan = self.scans[ref['scan_id']] ,尽管有方法可以改写dict为两组np.array,但是这个dict的value是一个自定义的对象!
class ScannetDataset(object):
def __init__(self, top_scan_dir, idx_to_semantic_cls_file,
instance_cls_to_semantic_cls_file, axis_alignment_info_file):
self.top_scan_dir = top_scan_dir
self.idx_to_semantic_cls_dict = read_dict(idx_to_semantic_cls_file)
self.semantic_cls_to_idx_dict = invert_dictionary(self.idx_to_semantic_cls_dict)
self.instance_cls_to_semantic_cls_dict = read_dict(instance_cls_to_semantic_cls_file)
self.semantic_cls_to_instance_cls_dict = defaultdict(list)
for k, v in self.instance_cls_to_semantic_cls_dict.items():
self.semantic_cls_to_instance_cls_dict[v].append(k)
self.scans_axis_alignment_matrices = read_dict(axis_alignment_info_file)这就很难办了:经过搜寻,我没有发现任何能在多进程中共享Python自定义对象的方法,这也启示我们,在写新任务的IO逻辑时,尽量设计numpy-compatible的数据结构!
更进一步:单机多实验时避免每次实验重新加载数据集
不少人应该也有这样的需求:grid-search时需要在单机的不同卡上跑不同超参的实验,但通用的IO方案很容易出现IO瓶颈,我们希望在所有实验中共享读取进内存的数据。
第零步:修改/dev/shm挂载的分区大小
默认的/dev/shm大小可能不满足需求,通过以下命令修改,重启后失效:
sudo mount -o size=60000M -o remount /dev/shm第一步:将数据整理为numpy格式
图片、文本、音频都很好通过预处理方式统一为向量形式。
第二步:Cache,将数据用memmap形式载入/dev/shm
memory_map可以将存储在磁盘上的内容映射到内存,考虑到我们有多个独立的主进程和无数DataLoader Worker需要读取数据进行IO,memory_map是一个很好的solution。Numpy提供了np.lib.format.open_memmap函数可以直接读取np.save的二进制文件,十分方便。同时考虑到读取效率,直接将数据cache到/dev/shm这个直接映射到内存的目录。
如果只是读,没有对mmap中数据的写操作,只需要两行code:
cache:
np.save(fn, array, allow_pickle=False)- fn是/dev/shm中某个文件的file handler,命名规则可以自定义。
- array是需要存储的数据,同上,请确认这个array的dtype不是object,否则allow_pickle不能为False,意味着之后的np.load需要重新反序列化,每个进程还是要加载一次这个object。
其他worker的读取:
np.lib.format.open_memmap(filename, mode='r')- 返回值等同于
np.load的结果,只不过内存开销极少,相当于创建了一个memory的view。
- 返回值等同于
如果涉及到读写,可能需要用到下面这个库,提供了对mmap的锁机制。
sharearray.py中的cache方法提供了读写的功能,建议使用。但不推荐多个独立进程读写mmap,毕竟没有人帮你测试这个锁机制是否完善...
一个demo供参考
"""
Each time after restarting the OS:
sudo mount -o size=60000M -o remount /dev/shm
"""
import os
import sys
sys.path.append(os.getcwd())
import argparse
from utils import share_array
import multiprocessing as mp
import numpy as np
from tqdm import tqdm
import time
SCENE_LEN = 1513
CACHE_PATH = None
CACHE_FILE_PREFIX = 'sharearray_'
KEYS = None
def _check(res_list):
res_set = set()
for k in KEYS:
res_set.add(len([p for p in res_list if p.endswith(k + '.npy')]))
return len(res_set) == 1 and list(res_set)[0] == SCENE_LEN
def get_workers(x):
if x == -1:
x = mp.cpu_count()
return x
def get_npy_list(npy_path):
res = [os.path.join(npy_path, p) for p in os.listdir(npy_path) if p.endswith('.npy')]
_check(res) # some simple check
return res
def cache_array(scene_path):
scene_name = os.path.basename(scene_path)[:-4] # scene0685_01_clip_region_feat
share_array.cache(scene_name, lambda: np.load(scene_path, allow_pickle=False),
shm_path=CACHE_PATH,
prefix=CACHE_FILE_PREFIX)
def clean_up_cache():
del_list = [os.path.join(CACHE_PATH, p) for p in os.listdir(CACHE_PATH) if os.path.basename(p).startswith(CACHE_FILE_PREFIX)]
for file in del_list:
try:
os.remove(file)
except FileNotFoundError:
print("Not Found: {}".format(file))
print('Clean up Done!')
if __name__ == '__main__':
parser = argparse.ArgumentParser()
parser.add_argument('--offline-2d-feat', type=str,
default='/data/meta-ScanNet/split_feat/',
help='The disk path for offline 2D features')
parser.add_argument('--cache-path', type=str, default='/dev/shm',
help='Cache target path, best be `/dev/shm` or other high-speed mount point (SSD, etc.)')
parser.add_argument('--workers', type=int, default=-1)
parser.add_argument('--debug', action='store_true', default=False)
parser.add_argument('--keys', nargs='+', default=['clip_region_feat', 'clip_scaled_region_feat', 'obj_feat',
'obj_coord', 'camera_pose', 'instance_id'])
args = parser.parse_args()
num_workers = get_workers(args.workers)
CACHE_PATH = args.cache_path
KEYS = args.keys
try:
scenes_list = get_npy_list(args.offline_2d_feat)
if not args.debug:
with mp.Pool(num_workers) as p:
_ = list(tqdm(p.imap(cache_array, scenes_list), total=len(scenes_list)))
# automatic join & close
else:
for scene_path in tqdm(scenes_list):
cache_array(scene_path)
print("Done Caching! Happy Training! Ctrl+C to clean up cache.")
while True: # ugly infinite loop
try:
time.sleep(1e9)
except KeyboardInterrupt:
break
print('Done!')
finally:
print('Cleaning up cache...')
clean_up_cache()
参考
Possible to share in-memory data between 2 separate processes?
